Aspartataminotransferas

Aspartataminotransferas (ASAT) (engelska: Aspartate transaminase (AST)) även känt som AspAT/ASAT/AAT eller (serum) glutaminoxaloättiksyratransaminas (GOT, SGOT), är ett pyridoxalfosfat (PLP)-beroende transaminasenzym (EC 2.6.1.1. beskrivet av Arthur Karmen et al. 1954.[1][2][3] ASAT katalyserar överföringen av en α-aminogrupp från aspartat till en ketosyra. ASAT förekommer främst i hjärta, lever och skelettmuskulatur, njurarna, hjärnan, röda blodkroppar och gallblåsan. I levern finns ASAT både i mitokondrier och i cytoplasman till skillnad från ALAT som bara finns i cytoplasman. Vid skador på något av dessa organ kan ASAT mätas i blodet. Mängden ASAT halveras i blodet på ungefär 17 timmar och i genomsnitt 87 timmar för mitokondriell ASAT.[4] Aminotransferas elimineras av sinusformade celler i levern.[4]
Funktioner
[redigera | redigera wikitext]Aspartattransaminas katalyserar omvandlingen av aspartat och α-ketoglutarat till oxaloacetat och glutamat.
- L-aspartat (Asp) + α-ketoglutarat ⇌ oxaloacetat + L-glutamat (Glu)
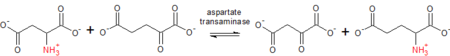
Som ett prototypiskt transaminas förlitar AST sig på PLP (vitamin B6) som en kofaktor för att överföra aminogruppen från aspartat eller glutamat till motsvarande ketosyra. I processen pendlar kofaktorn mellan PLP och pyridoxaminfosfatformen (PMP).[5] Aminogruppöverföringen som katalyseras av detta enzym är avgörande i både aminosyranedbrytning och biosyntes. Vid aminosyranedbrytning, efter omvandlingen av α-ketoglutarat till glutamat, genomgår glutamat därefter oxidativ deaminering för att bilda ammoniumjoner, som utsöndras som urea. I den omvända reaktionen kan aspartat syntetiseras från oxaloacetat, som är en nyckelmellanprodukt i citronsyracykeln.[6]
Isoenzymer
[redigera | redigera wikitext]Två isoenzymer finns i en mängd olika eukaryoter. Hos människor:
- GOT1 /cAST, det cytosoliska isoenzymet härrör huvudsakligen från röda blodkroppar och hjärta.
- GOT2 /mAST, mitokondriella isoenzymet finns övervägande i levern.
Dessa isoenzymer tros ha utvecklats från en vanlig förfäders-AST via genduplicering, och de delar en sekvenshomologi på cirka 45 procent.[7]
AST har också hittats i ett antal mikroorganismer, som E. coli, H. mediterranei,[8] och T. thermophilus.[9] I E. coli kodas enzymet av aspC-genen och har också visat sig visa aktiviteten hos en aromatisk aminosyratransaminas (EC 2.6.1.57).[10]
Struktur
[redigera | redigera wikitext]
Röntgenkristallografistudier har utförts för att bestämma strukturen av aspartattransaminas från olika källor, som kycklingmitokondrier,[11] grishjärta cytosol,[12] och E. coli.[13][14] Sammantaget är den tredimensionella polypeptidstrukturen för alla arter ganska lika. AST är dimer, bestående av två identiska underenheter, var och en med cirka 400 aminosyrarester och en molekylvikt av cirka 45 kD.[7] Varje underenhet är sammansatt av en stor och en liten domän, samt en tredje domän som består av de N-terminala resterna 3-14. Dessa få rester bildar en sträng som länkar och stabiliserar de två underenheterna av dimeren. Den stora domänen, som utgörs av resterna 48-325, binder PLP-kofaktorn via en aldiminkoppling till e-aminogruppen i Lys258. Andra rester i denna domän – Asp 222 och Tyr 225 – interagerar också med PLP via vätebindning. Den lilla domänen består av resterna 15-47 och 326-410 och representerar en flexibel region som skiftar enzymet från en "öppen" till en "stängd" konformation vid substratbindning.[11][14][15]
De två oberoende aktiva platserna är placerade nära gränssnittet mellan de två domänerna. Inom varje aktiv plats är ett par argininrester ansvariga för enzymets specificitet för dikarboxylsyrasubstrat där Arg386 interagerar med substratets proximala (α-)karboxylatgrupp, medan Arg292 komplexbinder med det distala (sidokedjan) karboxylatet.[11][14]
När det gäller sekundär struktur innehåller AST både α- och β-element. Varje domän har ett centralt ark av β-strängar med α-helixar packade på vardera sidan.
Mekanism
[redigera | redigera wikitext]Aspartattransaminas, som med alla transaminaser, fungerar genom dubbel substratigenkänning. Det vill säga att den kan känna igen och selektivt binda två aminosyror (Asp och Glu) med olika sidokedjor.[16] I båda fallen består transaminasreaktionen av två liknande halvreaktioner som utgör vad som kallas en ping-pong-mekanism. I den första halvreaktionen reagerar aminosyra 1 (till exempel L-Asp) med enzym-PLP-komplexet för att generera ketosyra 1 (oxaloacetat) och det modifierade enzymet-PMP. I den andra halvreaktionen reagerar ketosyra 2 (α-ketoglutarat) med enzym-PMP för att producera aminosyra 2 (L-Glu), vilket regenererar det ursprungliga enzymet-PLP i processen. Bildning av en racemisk produkt (D-Glu) är mycket sällsynt.[17]
De specifika stegen för halvreaktionen av Enzyme-PLP + aspartat ⇌ Enzym-PMP + oxaloacetat är följande (se figur); den andra halvreaktionen (ej visad) fortskrider på omvänt sätt, med α-ketoglutarat som substrat.[5][6]

- Intern aldiminbildning: Först bildar e-aminogruppen i Lys258 en Schiff-basbindning med aldehydkolet för att generera en intern aldimin.
- Transaldiminering: Den inre aldiminen blir sedan en extern aldimin när ε-aminogruppen i Lys258 ersätts av aminogruppen i aspartat. Denna transaldiminationsreaktion sker via en nukleofil attack av den deprotonerade aminogruppen av Asp och fortsätter genom en tetraedrisk mellanprodukt. Som denna punkt stabiliseras karboxylatgrupperna i Asp av guanidiniumgrupperna i enzymets Arg386- och Arg 292-rester.
- Kinonoidbildning: Vätet som är fäst till a-kolet i Asp abstraheras sedan (Lys258 tros vara protonacceptorn) för att bilda en kinonoidmellanprodukt.
- Ketiminbildning: Kinonoiden reprotoneras, men nu vid aldehydkolet, för att bilda ketiminmellanprodukten.
- Ketiminhydrolys: Slutligen hydrolyseras ketiminen för att bilda PMP och oxaloacetat.
Denna mekanism tros ha flera delvis hastighetsbestämmande steg.[18] Det har dock visats att substratbindningssteget (transaldimination) driver den katalytiska reaktionen framåt.[19]
Klinisk betydelse
[redigera | redigera wikitext]AST liknar alanintransaminas (ALT) genom att båda enzymerna är förknippade med leverparenkymceller. Skillnaden är att ALAT finns övervägande i levern, med kliniskt försumbara mängder i njurarna, hjärtat och skelettmuskulaturen, medan AST finns i levern, hjärtat (hjärtmuskeln), skelettmuskulaturen, njurarna, hjärnan och röda blodkroppar. Som ett resultat är ALAT en mer specifik indikator på leverinflammation än ASAT, eftersom ASAT kan vara förhöjt även vid sjukdomar som påverkar andra organ, såsom hjärtinfarkt, akut pankreatit, akut hemolytisk anemi, svåra brännskador, akut njursjukdom, muskel- och skelettsjukdomar och trauma.[20]
AST definierades 1954 som en biokemisk markör för diagnos av akut hjärtinfarkt. Användningen av AST för en sådan diagnos är dock nu överflödig och har ersatts av hjärttroponinerna.[21]
Laboratorietester ska alltid tolkas med referensintervallet från det laboratorium som utförde testet. Exempel på referensintervall visas nedan:
| Patienttyp | Referensintervall[22] |
| Man | 8 - 40 IE/l |
| Kvinna | 6 - 34 IE/l |
Se även
[redigera | redigera wikitext]- Levervärden
- Alaninaminotransferas (ALT/ALAT/SGPT)
- Transamimnaser
Referenser
[redigera | redigera wikitext]- Den här artikeln är helt eller delvis baserad på material från engelskspråkiga Wikipedia, Aspartate transaminase, 22 juli 2023.
Noter
[redigera | redigera wikitext]- ^ ”Transaminase activity in human blood”. The Journal of Clinical Investigation 34 (1): sid. 126–131. January 1955. doi:. PMID 13221663.
- ^ ”A note on the spectrometric assay of glutamic-oxalacetic transaminase in human blood serum”. The Journal of Clinical Investigation 34 (1): sid. 131–133. January 1955. doi:. PMID 13221664.
- ^ ”Serum glutamic oxaloacetic transaminase activity in human acute transmural myocardial infarction”. Science 120 (3117): sid. 497–499. September 1954. doi:. PMID 13195683. Bibcode: 1954Sci...120..497L.
- ^ [a b] ”Liver enzyme alteration: a guide for clinicians”. CMAJ 172 (3): sid. 367–379. February 2005. doi:. PMID 15684121. ”Aminotransferase clearance is carried out within the liver by sinusoidal cells. The half-life in the circulation is about 47 hours for ALT, about 17 hours for total AST and, on average, 87 hours for mitochondrial AST.”.
- ^ [a b] ”Mechanism of action of aspartate aminotransferase proposed on the basis of its spatial structure”. Journal of Molecular Biology 174 (3): sid. 497–525. April 1984. doi:. PMID 6143829.
- ^ [a b] Biochemistry. W.H. Freeman. 2006. Sid. 656–660. ISBN 978-0-7167-8724-2.
- ^ [a b] ”Recent topics in pyridoxal 5'-phosphate enzyme studies”. Annual Review of Biochemistry 59: sid. 87–110. 1990. doi:. PMID 2197992.
- ^ ”Purification and characterization of aspartate aminotransferase from the halophile archaebacterium Haloferax mediterranei”. The Biochemical Journal 278 (1): sid. 149–154. August 1991. doi:. PMID 1909112.
- ^ ”An aspartate aminotransferase from an extremely thermophilic bacterium, Thermus thermophilus HB8”. Journal of Biochemistry 119 (1): sid. 135–144. January 1996. doi:. PMID 8907187.
- ^ ”Escherichia coli mutants deficient in the aspartate and aromatic amino acid aminotransferases”. Journal of Bacteriology 130 (1): sid. 429–440. April 1977. doi:. PMID 15983.
- ^ [a b c] ”X-ray structure refinement and comparison of three forms of mitochondrial aspartate aminotransferase”. Journal of Molecular Biology 225 (2): sid. 495–517. May 1992. doi:. PMID 1593633.
- ^ ”Refinement and comparisons of the crystal structures of pig cytosolic aspartate aminotransferase and its complex with 2-methylaspartate”. The Journal of Biological Chemistry 272 (28): sid. 17293–17302. July 1997. doi:. PMID 9211866.
- ^ ”Three-dimensional structure of aspartate aminotransferase from Escherichia coli at 2.8 A resolution”. Journal of Biochemistry 104 (3): sid. 317–318. September 1988. doi:. PMID 3071527.
- ^ [a b c] ”Activity and structure of the active-site mutants R386Y and R386F of Escherichia coli aspartate aminotransferase”. Biochemistry 30 (7): sid. 1980–1985. February 1991. doi:. PMID 1993208.
- ^ ”Domain closure in mitochondrial aspartate aminotransferase”. Journal of Molecular Biology 227 (1): sid. 197–213. September 1992. doi:. PMID 1522585.
- ^ ”Dual substrate recognition of aminotransferases”. Chemical Record 5 (3): sid. 160–172. 2005. doi:. PMID 15889412.
- ^ ”Mechanism of racemization of amino acids by aspartate aminotransferase”. European Journal of Biochemistry 203 (3): sid. 563–569. February 1992. doi:. PMID 1735441.
- ^ ”The reaction catalyzed by Escherichia coli aspartate aminotransferase has multiple partially rate-determining steps, while that catalyzed by the Y225F mutant is dominated by ketimine hydrolysis”. Biochemistry 35 (16): sid. 5280–5291. April 1996. doi:. PMID 8611515.
- ^ ”Conformational change in aspartate aminotransferase on substrate binding induces strain in the catalytic group and enhances catalysis”. The Journal of Biological Chemistry 278 (11): sid. 9481–9488. March 2003. doi:. PMID 12488449.
- ^ ”AST/ALT”. www.rnceus.com. http://www.rnceus.com/lf/lfast.html.
- ^ ”The role of existing and novel cardiac biomarkers for cardioprotection”. Current Opinion in Investigational Drugs 8 (9): sid. 711–717. September 2007. PMID 17729182.
- ^ ”Alanine aminotransferase: analyte monograph”. Association for Clinical Biochemistry and Laboratory Medicine. 2012. 3–7. http://www.acb.org.uk/Nat%20Lab%20Med%20Hbk/ALT.pdf.
Vidare läsning
[redigera | redigera wikitext]- ”Structural basis for catalysis by aspartate aminotransferase”. Biological Macromolecules and Assemblies. "3". New York: Wiley. 1987. Sid. 187–285. ISBN 978-0-471-85142-4.
- ”Aspartate aminotransferase of Escherichia coli: nucleotide sequence of the aspC gene”. Journal of Biochemistry 97 (4): sid. 1259–1262. April 1985. doi:. PMID 3897210.
- ”The complete amino acid sequence of aspartate aminotransferase from Escherichia coli: sequence comparison with pig isoenzymes”. Biochemical and Biophysical Research Communications 122 (1): sid. 62–67. July 1984. doi:. PMID 6378205.
- ”Site-directed mutagenesis of Escherichia coli aspartate aminotransferase: role of Tyr70 in the catalytic processes”. Biochemistry 30 (31): sid. 7796–7801. August 1991. doi:. PMID 1868057.
Externa länkar
[redigera | redigera wikitext] Wikimedia Commons har media som rör Aspartataminotransferas.
Wikimedia Commons har media som rör Aspartataminotransferas.- AST - Lab Tests Online
- AST: MedlinePlus Medical Encyclopedia
| |||||
